Etiology
Os orthoebolavirus pertencem à família Filoviridae (gênero Orthoebolavirus; ordem: Mononegavirales). Esses vírus são estruturas alongadas e filamentosas de comprimento variável.[Figure caption and citation for the preceding image starts]: Eletromicrografia de transmissão mostrando parte da morfologia ultraestrutural apresentada por um vírion do vírus EbolaCentros de Controle e Prevenção de Doenças [Citation ends].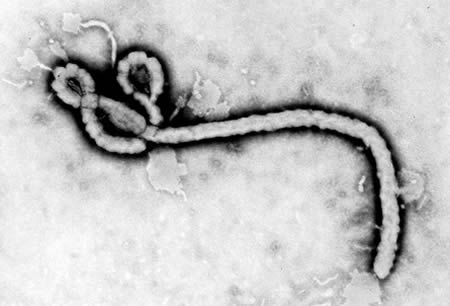
Acredita-se que o vírus seja inicialmente contraído a partir da exposição a fluidos corporais ou ao tecido de animais infectados, como morcegos e primatas não humanos; no entanto, o hospedeiro natural e o modo de transmissão para humanos ainda não foram confirmados.[39] Exames laboratoriais de competência de hospedeiros mostram que a infecção é possível em morcegos e roedores, mas não em plantas ou artrópodes.[40][41][42] A transmissão de animais para seres humanos pode ocorrer durante a caça e o consumo de espécies hospedeiras ou primatas não humanos infectados. Acredita-se, também, que a prática local de comer carne de animais selvagens ou alimento contaminado com fezes de morcego (3 espécies de morcego que se abrigam em árvores forem implicados como hospedeiros) contribua.
[Figure caption and citation for the preceding image starts]: Ecologia do vírus Ebola mostrando os ciclos enzoótico e epizoóticoCentros de Controle e Prevenção de Doenças [Citation ends].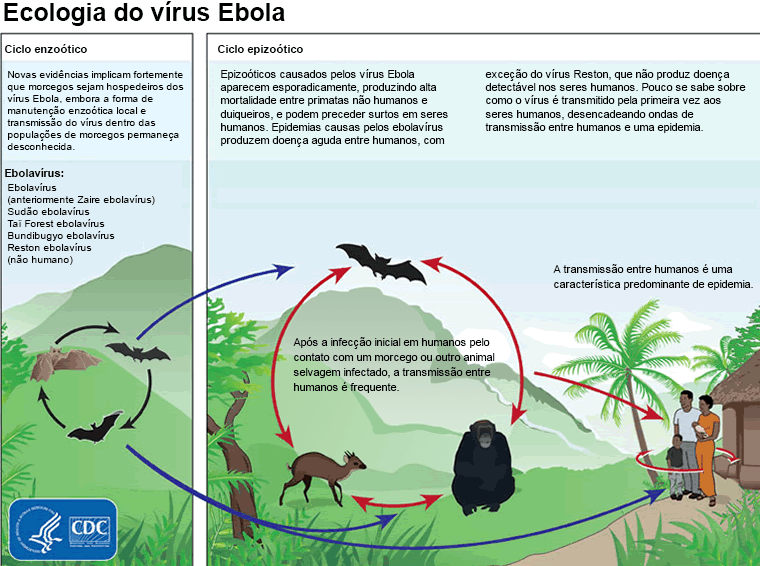
A transmissão entre humanos ocorre pelo contato direto com fluidos corporais de pacientes infectados ou objetos contaminados com fluido corporal infectado.[43][44] Na epidemia inicial, a reutilização de injeções não esterilizadas foi responsável por muitas das transmissões associadas aos cuidados de saúde.[24] No entanto, embora isso ainda represente um risco, a maioria dos casos resulta de contato físico próximo ou contato com fluidos corporais (por exemplo, suor, sangue, fezes, vômito, saliva, secreções genitais [incluindo sêmen], líquido amniótico e leite materno) de pacientes infectados.
Os níveis de vírus no sangue aumentam à medida que a doença evolui, e os pacientes são mais infecciosos em estágios avançados da doença (ou seja, quando ocorrem diarreia, vômitos e hemorragia).[45] Grandes quantidades de vírus podem ser encontradas na pele, e tocar um paciente infectado pode resultar em transmissão, pois o suor também pode conter o vírus.[46]
Eventos de superdisseminação na comunidade também são cada vez mais reconhecidos como fatores contribuintes; o funeral de um tradicional curandeiro em Serra Leoa, ocorrido em 2015, foi relacionado a 300 casos.[47] Em um estudo, descobriu-se que superdisseminadores foram responsáveis por aproximadamente 61% das infecções no surto de 2014.[48]
Em um estudo para identificar a taxa de eliminação de partículas virais em vários fluidos corporais, o vírus foi isolado a partir da saliva, do leite materno, das fezes, das lágrimas e do sêmen até 40 dias após o início da doença.[44][49][50] O vírus ainda pode ser detectado no sêmen mais de 12 meses após a recuperação da infecção, possivelmente porque o tecido testicular seja um local protegido contra o sistema imunológico.[51] Isso significa que a transmissão sexual talvez seja possível muito depois de a infecção ter sido resolvida, e esses casos foram confirmados durante e após o surto de 2014.[44][49][50][52][53][54][55][56] O vírus foi também detectado no fluido vaginal.[57] Os outros sítios imunologicamente protegidos incluem o interior dos olhos, a placenta e o sistema nervoso central, particularmente o líquido cefalorraquidiano.[58] Partículas virais podem continuar a ser eliminadas através da urina e do suor. Em um paciente que se recuperou na Alemanha, o vírus foi detectado na urina 14 dias após a negativação sorológica e, no suor, por até 19 dias após a negativação sorológica.[46]
Demonstrou-se que a infecção por via inalatória é possível em primatas não humanos; porém, não há evidências de transmissão pelo ar em seres humanos.[14][59] Ainda se deve considerar a possibilidade de transmissão oportunista do vírus através do ar durante vômitos forçados (semelhante àquela observada com a infecção por norovírus) e durante procedimentos geradores de aerossóis associados a intervenções de cuidados intensivos.
Fora das áreas endêmicas, a doença do Ebola é rara e, em geral, uma infecção importada.[60] Viajantes que chegam de áreas afetadas, bem como cientistas e outras pessoas que trabalham com materiais e animais possivelmente infectados, estão em alto risco.
პათოფიზიოლოგია
Tem havido avanços importantes na elucidação da patogênese da infecção; no entanto, a maioria dos estudos tem sido realizada em primatas não humanos e modelos de roedores.[7] Isso se deve às dificuldades em conduzir estudos com seres humanos em contextos com poucos recursos onde essas infecções ocorrem naturalmente.
O genoma do vírus consiste em uma fita simples de 19 kb de ácido ribonucleico (RNA) de sentido negativo com 7 genes virais transcritos pela polimerase de RNA dependente de RNA presente no vírion. A fita simples do RNA é coberta por nucleoproteínas virais NP e VP30 organizadas em hélice que estão ligadas pelas proteínas da matriz VP24 e VP4 à bicamada lipídica que reveste o vírion.[14] Houve uma rápida mutação do vírus no surto de 2014, levantando preocupações quanto a sua capacidade de escapar das respostas imunes do hospedeiro e evoluir sob pressão de novas terapias.[61][62]
O período de incubação após a infecção é de 2 a 21 dias.[3] O número reprodutivo (R₀) básico, o número de casos secundários gerados por um caso primário em uma população totalmente suscetível, foi estimado em 1.3 a 2. No entanto, o R₀ varia significativamente entre os países e os surtos. A R₀ média geral combinada foi estimada em 1.95, mas já chegou a 10 em alguns surtos. Em países onde os casos são importados, o valor variou de 1.5, nos EUA, a 1.6, no Reino Unido, e 2.4, em Gâmbia.[63]
A invasão do tecido ocorre através de fluido infectado que entra em contato com rupturas na mucosa ou pele. Isso pode ocorrer com a transmissão de animais para seres humanos ou entre humanos. Monócitos, macrófagos e células dendríticas são os locais de replicação preferidos pelos filovírus na infecção inicial. As células infectadas migram para os linfonodos regionais, o fígado e o baço, disseminando assim a infecção. O vírus Ebola tem amplo tropismo celular e é capaz de infectar diversos tipos de células, mas a replicação viral extensiva ocorre no tecido linfoide, fígado e baço.[7][14][64] Ele também tem a notável capacidade de modular a expressão dos genes envolvidos na resposta imune do hospedeiro, causando apoptose dos linfócitos e atenuação dos efeitos protetores da interferona.[65][66][67][68][69]
A resposta imune do hospedeiro é crucial e determina o desfecho da infecção. A evolução para o estágio grave do espectro da doença ocorre quando o vírus desencadeia a expressão de uma variedade de citocinas pró-inflamatórias, entre elas: interferonas; interleucinas (IL) como IL-2, IL-6, IL-8 e IL-10; proteína induzível pela interferona; e fator de necrose tumoral alfa (TNF).[7][14][70] Isso, por sua vez, causa ativação endotelial e redução da integridade vascular, liberação do fator tecidual (com início associado de coagulopatia) e aumento dos níveis de óxido nítrico (com hipotensão associada).[71] A infecção causa depleção linfocítica por meio de apoptose indireta (uma vez que o vírus não se replica em linfócitos) e supressão de neutrófilos via glicoproteína (GP).[72] A causa mais comum de trombocitopenia é o desaparecimento das plaquetas do tecido danificado ou a coagulação intravascular disseminada mais generalizada induzida pelo vírus, situação em que há depleção dos fatores de coagulação.[73] A coagulação intravascular disseminada, junto com comprometimento hepático agudo, predispõe o paciente a complicações por sangramento. Outras complicações da doença grave incluem lesão renal aguda, hepatite e pancreatite.[14] A resposta precoce dos anticorpos, juntamente com a redução da depleção linfocítica, está associada ao clearance viral efetivo e à sobrevida.[74] A citometria de fluxo, que foi usada em um centro de tratamento na Guiné durante o surto de 2014, demonstrou que a desregulação das células T (caracterizada por uma expressão maior de CTLA-4 e PD-1 nas células CD4 e CD8) estava associada à morte. Isso confirma sugestões anteriores de que uma resposta imune adequada, mas controlada, é essencial para a sobrevida.[75]
O desenvolvimento de choque ainda não é bem compreendido. Múltiplos fatores podem contribuir, entre eles: sepse bacteriana, possivelmente por meio da translocação da bactéria através do intestino; um efeito direto do vírus; coagulação intravascular disseminada; ou hemorragia.[70]
Classificação
Taxonomia do vírus
Os orthoebolavirus pertencem à família Filoviridae (gênero Orthoebolavirus).[1]
Atualmente existem seis espécies conhecidas de Orthoebolavirus (observação: os nomes das espécies foram atualizados em abril de 2023):[2]
Vírus Ebola (EBOV) - Orthoebolavirus zairense (anteriormente conhecido como Zaire ebolavírus)
Vírus do Sudão (SUDV) - Orthoebolavirus sudanense (anteriormente conhecido como ebolavírus do Sudão)
Vírus de Tai Forest (TAFV) - Orthoebolavirus taiense (anteriormente conhecido como ebolavírus de Tai Forest ou ebolavírus da Costa do Marfim)
Vírus de Bundibugyo (BDBV) - Orthoebolavirus bundibugyoense (anteriormente conhecido como ebolavírus de Bundibugyo)
Vírus de Reston (RESTV) - Orthoebolavirus restonense (anteriormente conhecido como ebolavírus de Reston)
Vírus de Bombali (BOMV) - Orthoebolavirus bombaliense (anteriormente conhecido como ebolavírus de Bombali).
O vírus de Reston e o vírus de Bombali não são conhecidos como causadores da doença em humanos.[1]
As quatro espécies que causam doença em humanos causam síndromes clínicas ligeiramente diferentes, de gravidade variável, e apresentam uma taxa de letalidade relatada de 25% a 90% entre diferentes surtos (a taxa média é de aproximadamente 50%).[3] O vírus do Ebola e o vírus do Sudão são especialmente conhecidos por sua virulência; as outras espécies são consideradas menos virulentas.
Vírus Ebola (Orthoebolavirus zairense):
Isolado pela primeira vez em 1976 durante um surto no norte do Zaire (atualmente conhecido como República Democrática do Congo [RDC]).[1] Esta parece ser a mais virulenta das seis espécies, e tem a maior taxa de letalidade de todas as espécies.[4] Ele é responsável pelo maior surto que teve início na África Ocidental em 2014.
Vírus do Sudão (Orthoebolavirus sudanense):
Isolado pela primeira vez em 1976 durante um surto no sul do Sudão. Causa uma síndrome idêntica ao Orthoebolavirus zairense; no entanto, a taxa de letalidade é menor.[4]
Vírus de Tai Forest (Orthoebolavirus taiense):
Somente um caso foi documentado em 1994 em uma pesquisadora suíça que realizou uma autópsia em um chimpanzé morto no Parque Nacional de Tai na Costa do Marfim.[5] Ela se recuperou da fase febril da doença sem nenhuma complicação hemorrágica.
Vírus de Bundibugyo (Orthoebolavirus bundibugyoense):
Descoberto em 2007 durante um surto isolado no distrito de Bundibugyo no oeste de Uganda. O vírus isolado foi identificado como uma espécie distinta, distantemente relacionada ao Orthoebolavirus taiense.[6]
Vírus de Reston (Orthoebolavirus restonense):
Isolado pela primeira vez em Reston, na Virgínia, nos EUA em 1989, onde foi encontrado em macacos Cynomolgus importados das Filipinas. Descobriu-se que vários trabalhadores expostos a animais infectados apresentavam sorologia positiva, mas nenhum sintoma clínico. Desde então, o vírus também foi isolado de porcos nas Filipinas.[7][8]
Vírus de Bombali (Orthoebolavirus bombaliense):
Descoberto pela primeira vez em Serra Leoa em 2018 nos órgãos do morcego angolano de cauda livre (Mops condylurus) e do pequeno morcego de cauda livre (Chaerephon pumilus). Foi também identificado em morcegos no Quênia e na Guiné.[9][10] Não se sabe se esse vírus é patogênico em humanos.[11]
Outras infecções filovirais
A família Filoviridae de vírus inclui o orthoebolavirus, o orthomarburgvirus e o Cuevavirus, entre outros. O Orthomarburgvirus é o único outro membro desse grupo conhecido por causar infecção em seres humanos. Ele foi isolado de morcegos e causa uma síndrome parecida com a doença do Ebola. Vários surtos foram relatados, frequentemente relacionados à exposição a animais em minas ou cavernas.[2][12]
O uso deste conteúdo está sujeito ao nosso aviso legal
